“Genetiğin dijital olduğu fikri beni büyülüyor. Bir gen, bilgisayar bilgileri gibi uzun bir kodlanmış harf dizisidir. Modern biyoloji, bilgi teknolojisinin bir dalı haline geliyor.” demiş biyolog yazar Richard
Anlatacaklarıma başlamadan önce Moleküler Biyolojinin Merkez Dogması üzerine konuşmak istiyorum. Çünkü tek hücreli RNA dizileme verilerini anlamak için önce bu konuda biraz bilgi sahibi olmak gerektiğini düşünüyorum.
Moleküler Biyolojinin Merkez Dogması nedir?
Moleküler biyolojinin temel dogması, biyolojik bir sistem içindeki genetik bilgi akışının bir açıklamasıdır. Orijinal anlamı bu olmasa da sıklıkla “DNA RNA yapar ve RNA protein yapar” şeklinde ifade edilir. İlk olarak 1957’de Francis Crick tarafından belirtildi, daha sonra 1958’de yayınlandı.
- Bu dogma, DNA’nın tüm proteinlerimizi yapmak için gerekli bilgiyi içerdiğini ve RNA’nın bu bilgiyi ribozomlara taşıyan bir haberci olduğunu öne sürer.
- Ribozomlar, bilginin bir koddan işlevsel ürüne ‘çevrildiği’ hücrede fabrikalar görevi görür.
- DNA talimatlarının fonksiyonel ürüne dönüştürüldüğü sürece gen ekspresyonu denir.
- Gen ifadesinin iki temel aşaması vardır — transkripsiyon ve translasyon.
- Transkripsiyonda, her hücrenin DNA’sındaki bilgiler küçük, taşınabilir RNA mesajlarına dönüştürülür.
- Translasyon sırasında, bu mesajlar DNA’nın hücre çekirdeğindeki yerden belirli proteinleri yapmak için “okundukları” ribozomlara gider.
- Sonuç olarak kısaca dogma, hücrelerimizde en sık görülen bilgi modelinin şu şekilde olduğunu belirtir:
- Mevcut DNA’dan yeni DNA yapmak için DNA replikasyonu.
- DNA’dan yeni RNA yapmak için transkripsiyon.
- RNA’dan yeni proteinler yapmak için translasyon.
ScRNA-seq’in (single-cell RNA sequencing) arkasındaki büyük fikir nedir?
Moleküler biyolojinin temel dogmasının ne olduğunu az çok öğrendiğimize göre hadi ScRNA-seq hakkında konuşalım.
Paralel olarak yüz binlerce hücrenin transkriptomlarını (bir hücrede bulunan toplam RNA) profillemek için yüksek verimli bir sıralama tekniğidir. Moleküler biyolojinin ana dogmasına göre, gen ifadesi, DNA (gen) → RNA → Proteinden gelen genetik bilginin tek yönlü akışı olduğunu söyledik. scRNA-seq, söz konusu her hücre için bu bilgi akışını RNA seviyesinde yakalar.
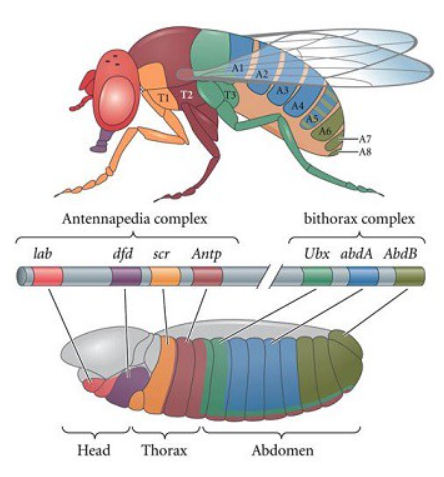
ScRNA-seq’in arkasındaki mantığı anlamak için, meyve sineği Drosophila’daki homeobox genlerine bir göz atalım. Homeobox terimi homeosis’ten gelir, yani bir vücut parçasının diğeriyle değiştirilmesi anlamına gelir.
Meyve sineğinde vücut örüntüsü homeotik gen kompleksleri antennapedia ve bithorax tarafından kontrol edilir. Bu genlerin larvadaki belirli bölgelerde ekspresyonu, yetişkin sineğin segment kimliklerini belirler.
Örneğin, Ubx geni (yukarıdaki resme bakınız), üçüncü torasik segmentte kanat oluşumunu engeller (T3, yukarıdaki şekle bakınız). Bu nedenle kanatlar sineğin T2 segmenti ile sınırlıdır.
Ubx geninin işlevsel bir kopyasına sahip olmayan bir ubx mutantında — T3, T2’ye dönüşerek mutant sineğe fazladan bir çift kanat verir! Drosophila gelişiminin büyüleyici yönleri hakkında daha fazlasını buradan okuyabilirsiniz.

Genel olarak, hücre kimlikleri, o hücrede başka bir hücrede bulunmayabilen (deri hücresine karşı nöron) özel genlerin ekspresyonu ile belirlenir. Gen etkileşim ağları, belirli bir genin ekspresyonunun başka bir genin ekspresyonunu tamamen durdurmasına neden olabileceği veya farklı bir genin ekspresyonunu etkinleştirebileceği vb. karmaşık doğrusal olmayan ağlardır (interaktom). Genlerin bu bağlamsal etkileşimi, bir hücrenin gelişimsel manzarasını belirler.

Ubx mutantında, bir kanat yapmak için neyin gerekli olduğunu anlamak için bir ubx mutantının T3 segmentinden RNA’yı normal bir sineğin T3 segmentiyle karşılaştırabiliriz. Çünkü gözlemlediğimiz fiziksel bir değişim (ekstra kanatlar ) gen ifadesinde temelleri olmalıdır. Her bir genden kopyalanan RNA kopyalarının sayısını sayarak (bir A / B test düzeneğindeki tıklamaları ve sayfa ziyaretlerini toplayabildiğimiz gibi), belirli bir mutasyonun gen ifadesindeki ince farklılıkları kesin olarak ölçebiliriz — bu çalışma diferansiyel gen ekspresyon analizi olarak bilinir. Diferansiyel gen ekspresyonu, scRNA-sekansının temelini oluşturur. Benzer şekilde, bir kanser hücresinin normal bir muadili ile karşılaştırılması, aksi takdirde kolayca tanımlanamayan benzersiz ilaç hedeflerini ortaya çıkarabilir.
Bu Nasıl Yapılır?
Veri biliminde, garbage in, garbage out (GIGO) iyi bilinen bir kavramdır. Burada analiz hattınıza hatalı verilerle beslerseniz, çıkardığınız şey çöp olur. Bununla başa çıkmanın tek yolu, öncelikle uygun bir rastgele deneysel tasarım ve anlamlı veri toplamaktır.
Tek Hücreli RNA Dizileme verilerinin elde edilmesi ve analizi adımları şu şekildedir:
- Örnek toplama: Analiz etmek için doku örnekleri toplanır.
- Tek hücrelerin dokulardan izolasyonu: Bu en zorlu kısımdır. Dokular bir hücre koleksiyonudur ve hassastır, bu nedenle dikkatli bir şekilde kullanılması gerekir. Daha fazla ayrıntıyı bu linkten bulabilirsiniz.

3. RNA’nın tek tek hücrelerden çıkarılması
4. RNA’yı DNA’ya dönüştürme: RNA’nın genomik kökenini belirlemek için DNA sıralanması gereklidir. Bu nedenle, dizilemeden önce, ters transkripsiyon polimeraz zincir reaksiyonu (RT-PCR) ile RNA’yı DNA’ya dönüştürmemiz gerekir. RNA, tek sarmallı bir nükleik asit molekülüdür, bu nedenle oldukça kararsız olan RT-PCR, RNA’yı dizileme deneyleri için uygun olan çok kararlı bir DNA’ya dönüştürür.
5. DNA Dizileme
6. Dizilim Analizi:
- Kalite Kontrol: Sıralama verilerinin kalitesi kontrol edilir. (fastQC, multiQC, rseqc)
- Adapter Trimming: Sıralama adaptörleri kaldırılır. (cutadapt, flexbar)
- Referans genom hizalaması: Bir sekansın kimliği belirlenir. (HISAT / HISAT2 gibi eklemeye duyarlı bir eşleyici tercih edilmelidir).
- Hizalama Görselleştirme: Hizalama dosyasın (IGV) görselleştirilir.
- Hizalama sonrası Kalite Kontrolü (QC): Hizalamadan sonra sıra kalitesi kontrol edilir (samtools, fastQC)
- Count Matrisi Hazırlanması: Dizileme analizi ile tespit edilen benzersiz RNA’ların sayısını sayılır ve kaydedilir. İşte hazırlanan bu matrix bizim kümeleme analizini yapacağımı veri setini oluşturuyor.
Dizilim analizi ile ilgili tutoriala bu linkte tıklayarak gidebilirsiniz.
Farkındayım buraya kadar çok fazla biyolojik terimler hakkında konuştuk. Umarım sabredip buraya kadar gelmişsinizdir. Analizini yapacağımız veri setinin nasıl elde edildiğini ve ne hakkında olduğunu serinin ilerleyen kısımlarında kafa karışıklığı olmaması adına açıklamaya çalıştım. Hadi şimdi veri analizi hakkında konuşalım.
7. Veri Analizi:
Benim bu projedeki görevim buradan itibaren başlıyor. Bundan önceki süreçlerde bulunmuyorum. Benim buradaki amacım elde edilen ScRNA-seq veri setini aşağıda anlatacağım çeşitli ön işlemelerden geçirdikten sonra denetimsiz (unsupervised) makine öğrenmesi yöntemlerinden olan kümeleme (cluster) algoritmalarını kullanarak veri setini kümerlere ayırmak. Hadi şimdi bu ön işleme ve kümeleme adımlarından bahsedelim.
- Hücre Kalite Kontrolü ve filtreleme: Tanımlanmış genlerin belirli bir eşiğinden az gen içeren hücrelerin çıkarılması.
- Gen Kalite Kontrolü ve filtreleme: Düşük kaliteye sahip genlerin çeşitli yöntemlerle saptanması ve elenmesi.
- Normalizasyon: Normalleştirmenin amacı, istenmeyen teknik varyasyonu ortadan kaldırmaktır
- Boyutsal İndirgeme: RNAseq verileri yüksek boyutlu verilerdir. Bu sebeple, PCA gibi çeşitli yöntemler kullanarak bu veri setinin boyutunu indirgememiz gerekmektedir.
- Kümeleme ve Görselleştime: Son olarak veri setini çeşitli algoritmalar kullanarak kümelere ayırmalı ve görselleştirmeliyiz.
Bu yazıda Tek Hücreli RNA Dizileme Verilerinin ne olduğundan, nasıl elde edildiklerinden ve kısaca veri analizi süreçlerinden bahsettik.
Bu yazı belki fazla teorik ve biyolojik terimler içerdi ama merak etmeyin serinin bundan sonraki bölümlerinde yukarıda anlattığım veri analizi kısmındaki adımları Python ve R programlama dilleri kullanarak yapacağı ve böylelikle kapsamlı bir şekilde Tek Hücreli RNA Dizileme verilerinin analizini yapmış olacağız.
Buraya kadar sabredip okuduğunuz için teşekkür ederim.
Kaynaklar: